
學(xué)術(shù)動態(tài)
劉慶友教授團(tuán)隊成功構(gòu)建山羊腸道微生物基因組目錄
發(fā)表時間:2023-10-13
2023年10月2日,Microbiome(Top 一區(qū),IF= 15.5)在線發(fā)表了題為“The multi-kingdom microbiome of the goat gastrointestinal tract”的學(xué)術(shù)論文。論文主要由我校生命科學(xué)與工程學(xué)院劉慶友教授團(tuán)隊、華中科技大學(xué)陳衛(wèi)華教授團(tuán)隊和廣西農(nóng)業(yè)職業(yè)技術(shù)大學(xué)曹艷紅研究員團(tuán)隊等共同完成,我校為第一署名單位,曹艷紅研究員、馮彤博士生和吳英健博士生為并列第一作者,劉慶友教授和華中科技大學(xué)陳衛(wèi)華教授為共同通訊作者。
山羊是世界范圍內(nèi)具有重要經(jīng)濟(jì)價值的家畜動物,通過提供肉類、奶制品和皮毛等產(chǎn)品,在人類生活中發(fā)揮著不可或缺的作用。據(jù)聯(lián)合國糧食及農(nóng)業(yè)組織(FAO)的數(shù)據(jù),全球約有1,000個山羊品種和超過10億只山羊,其中中國是世界上山羊數(shù)量最多的國家之一,約有1.4億只山羊。反芻動物的胃腸道微生態(tài)群落在其宿主的生理功能中具有重要作用。論文構(gòu)建了包含4,075個細(xì)菌和古細(xì)菌基因組裝(MAGs)以及7,204個病毒基因組的山羊微生物基因組目錄(GMMC),并注釋了4,817,256個非冗余蛋白編碼基因。填補(bǔ)了黑山羊腸道微生物研究中的空白,提供了涵蓋細(xì)菌、古細(xì)菌和病毒基因組和編碼蛋白的目錄,為實現(xiàn)更好的山羊營養(yǎng)代謝和健康養(yǎng)殖奠定了基礎(chǔ)。
劉慶友教授團(tuán)隊主要致力于多組學(xué)解析動物重要經(jīng)濟(jì)性狀形成的分子調(diào)控機(jī)制,為廣東省動物分子設(shè)計與精準(zhǔn)育種重點(diǎn)實驗室牛羊草食動物團(tuán)隊負(fù)責(zé)人、第四屆國家畜禽遺傳資源委員會牛專業(yè)委員會成員,第五屆全國畜牧學(xué)科高峰論壇副秘書長,國家自然科學(xué)基金聯(lián)合基金重點(diǎn)項目會評專家等。該團(tuán)隊已在《Nature Communications》《National Science Review》《Microbiome》《Molecular
Biology and Evolution》等知名國際學(xué)術(shù)期刊發(fā)表了系列論文。
結(jié)果解讀
1、山羊腸道微生物基因組目錄(GMMC)的構(gòu)建
團(tuán)隊共采集了497份樣本,包括十個山羊胃腸道位點(diǎn),胃(瘤胃、網(wǎng)胃、瓣胃、皺胃)、小腸(十二指腸、空腸、回腸)和大腸(盲腸、結(jié)腸),三個發(fā)育階段(1月齡、6月齡和12月齡;見表S3)、兩種喂養(yǎng)方式(圈養(yǎng)和放養(yǎng))和四個地理位置(中國的云南、四川、廣西和海南省)。獲得2.7 Tb的clean
data數(shù)據(jù),組裝了共計4,075個長度超過200kb的MAGs,包括4004個細(xì)菌MAGs和71個古細(xì)菌MAGs。我們還注釋7,204個非冗余病毒基因組,最小長度為5kb。我們將這4,004個細(xì)菌MAGs和71個古細(xì)菌MAGs以及7,204個病毒基因組稱為山羊多界微生物組目錄(GMMC),給出了更好地代表山羊胃腸道微生物群落的基因組/非冗余蛋白的序列參考藍(lán)圖,同時包含了相當(dāng)比例的新穎基因組作為待發(fā)掘的方向。
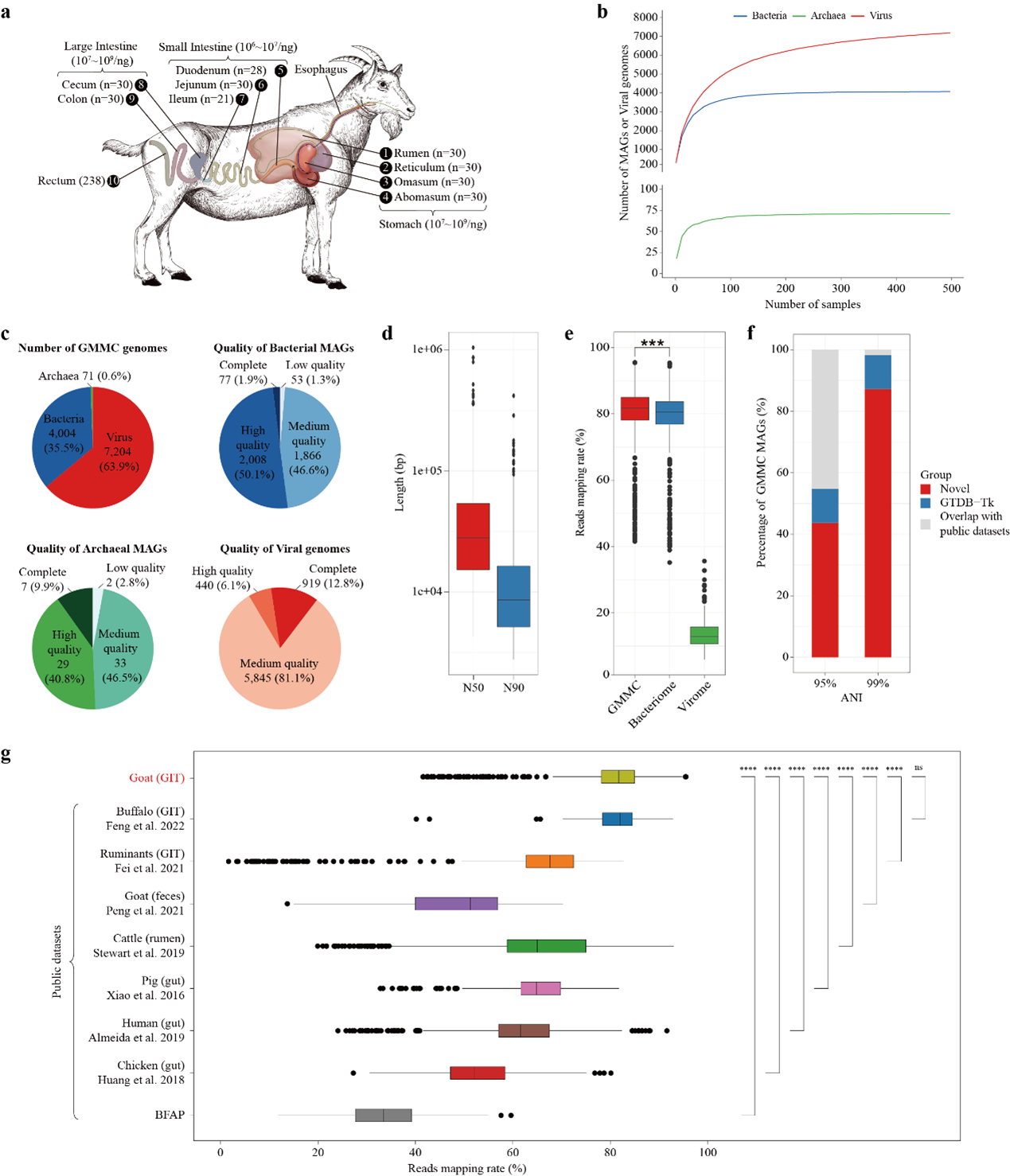
圖1 山羊胃腸道微生物基因組的構(gòu)建
2. GMMC的分類和功能注釋
在4,075個MAGs中,所有MAGs都能被分配到其各自的界(細(xì)菌,n = 4,004;古菌,n
= 71),大多數(shù)MAGs能夠在門、綱、目和科的層次上被分配到已知的分類,在種層面上,有451個MAGs(11.07%)能夠被分類為已知的物種,表明大多數(shù)MAGs以前尚未被鑒定。在門分類上,細(xì)菌MAGs主要由Firmicutes_A(n = 1,503)和Bacteroidota(n
= 1,479)作為優(yōu)勢菌門,其次是Verrucomicrobiota、Proteobacteria和Spirochaetota。所有的古細(xì)菌MAGs都是已知的產(chǎn)甲烷菌,屬于Thermoplasmatota門(n
= 37)、Halobacterota門(n = 30)和Euryarchaeota門(n
= 4)。我們使用VirusTaxo和Demovir工具對GMMC病毒基因組進(jìn)行了注釋。我們將其中的75.67%分配到已知的分類類群中;其中,602個病毒被分配到Poxviridae科,其次是Mimiviridae(n = 573)、Microviridae(n = 477)和Siphoviridae(n
= 120)。總體分類分布與其他來源于人類的宏基因組病毒目錄(包括GVD、MGV和GPD)類似。我們從所有的GMMC基因組中注釋了7,645,971個蛋白編碼基因,并使用CD-HIT在95%氨基酸相似性閾值下去冗余,得到了4,817,256個非冗余基因。
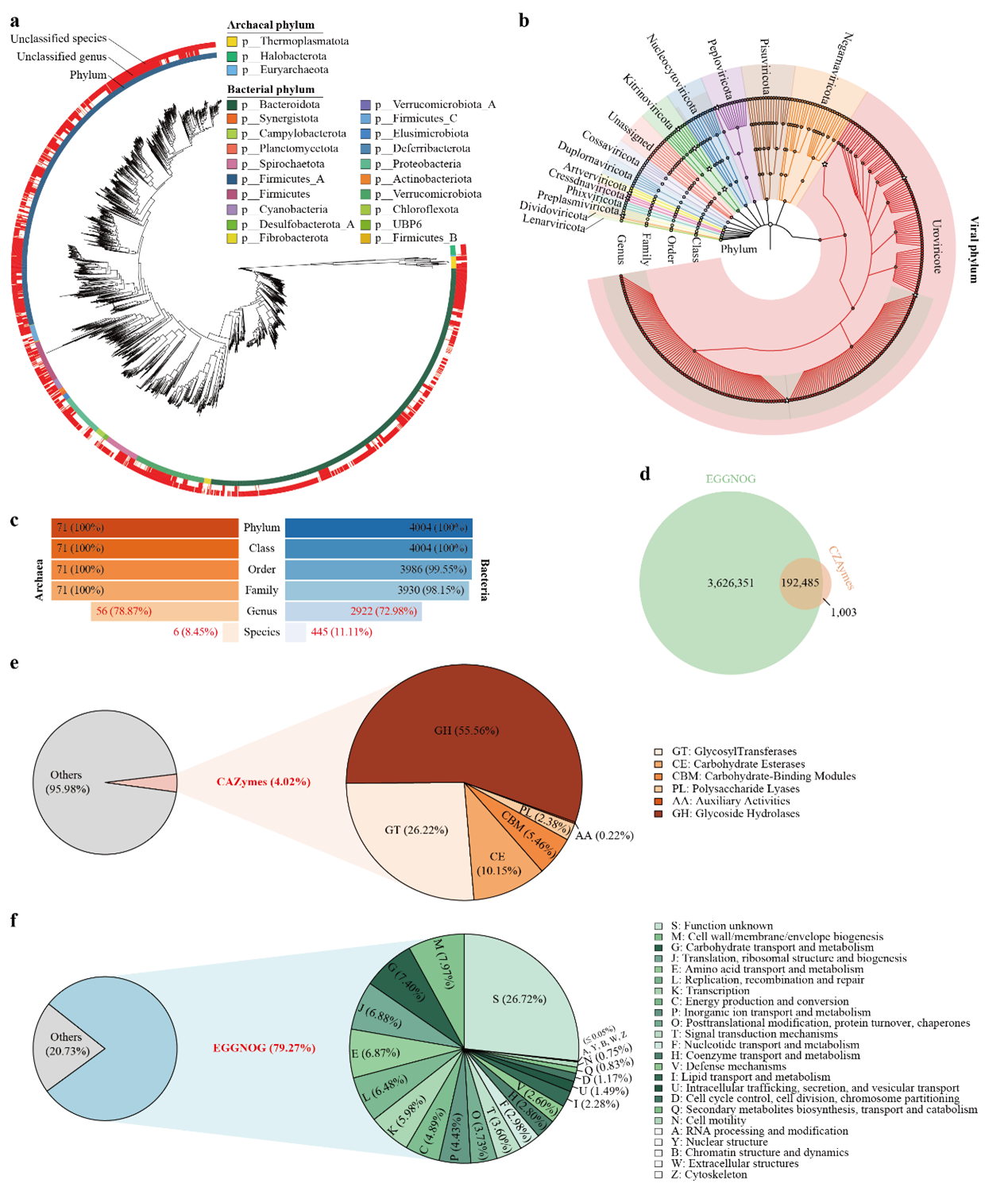
圖1 山羊胃腸道微生物基因組的構(gòu)建
2. GMMC的分類和功能注釋
在4,075個MAGs中,所有MAGs都能被分配到其各自的界(細(xì)菌,n = 4,004;古菌,n
= 71),大多數(shù)MAGs能夠在門、綱、目和科的層次上被分配到已知的分類,在種層面上,有451個MAGs(11.07%)能夠被分類為已知的物種,表明大多數(shù)MAGs以前尚未被鑒定。在門分類上,細(xì)菌MAGs主要由Firmicutes_A(n = 1,503)和Bacteroidota(n
= 1,479)作為優(yōu)勢菌門,其次是Verrucomicrobiota、Proteobacteria和Spirochaetota。所有的古細(xì)菌MAGs都是已知的產(chǎn)甲烷菌,屬于Thermoplasmatota門(n
= 37)、Halobacterota門(n = 30)和Euryarchaeota門(n
= 4)。我們使用VirusTaxo和Demovir工具對GMMC病毒基因組進(jìn)行了注釋。我們將其中的75.67%分配到已知的分類類群中;其中,602個病毒被分配到Poxviridae科,其次是Mimiviridae(n = 573)、Microviridae(n = 477)和Siphoviridae(n
= 120)。總體分類分布與其他來源于人類的宏基因組病毒目錄(包括GVD、MGV和GPD)類似。我們從所有的GMMC基因組中注釋了7,645,971個蛋白編碼基因,并使用CD-HIT在95%氨基酸相似性閾值下去冗余,得到了4,817,256個非冗余基因。
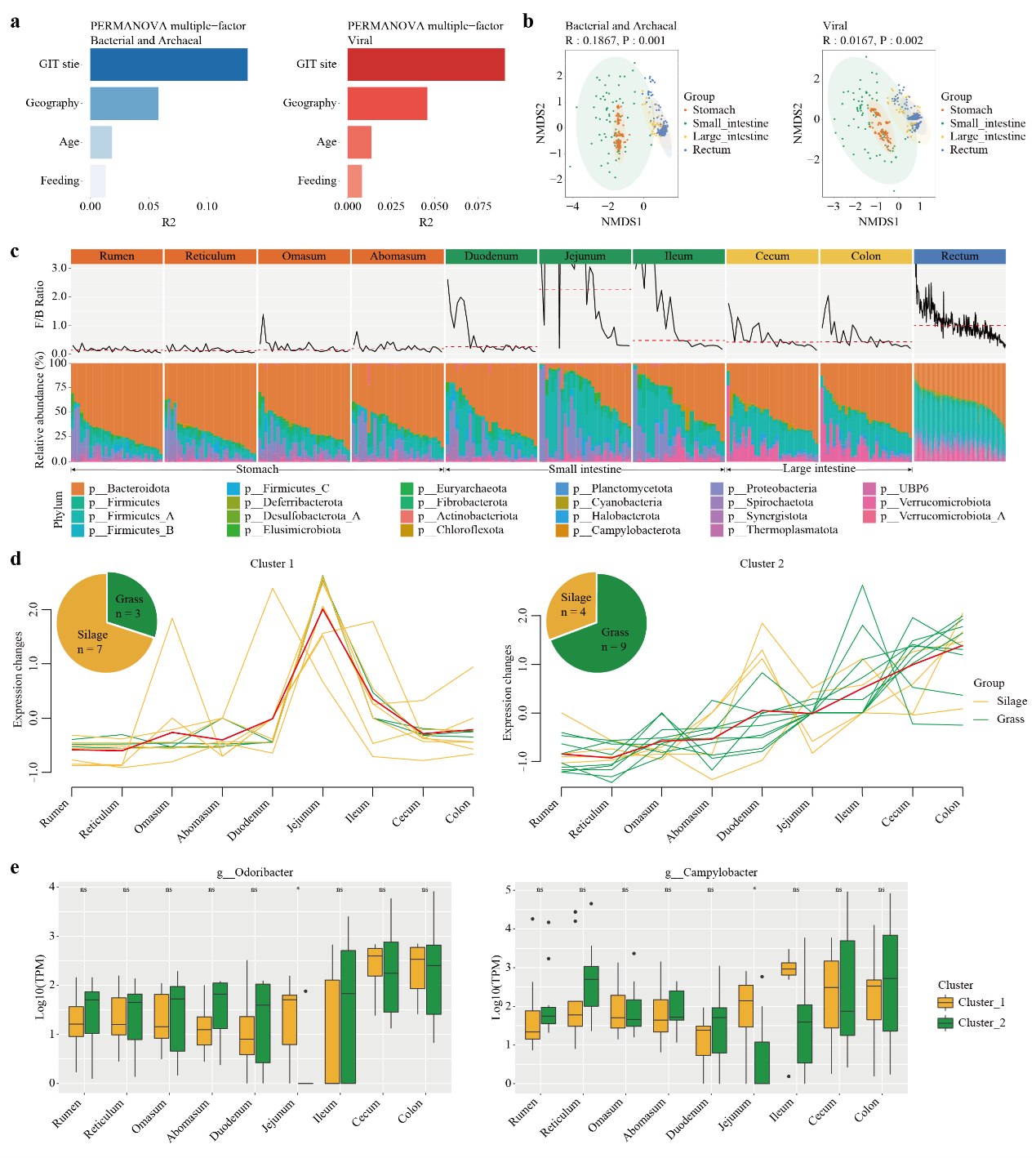
圖3 山羊胃腸道微生物群落動態(tài)變化及飲食影響
4. 山羊胃腸道微生物類群的分布和變化
論文觀察到注釋的纖維素消化細(xì)菌在胃腸道部位之間存在顯著變化,包括六個屬:Prevotella、Fibrobacter、Faecalibacterium、Acetivibrio_A、Ruminiclostridium_E和Paraprevotella。前兩個屬,即Prevotella和Fibrobacter在胃和小腸中的相對豐度較高,而其他四個屬在下游部位(即大腸和直腸)中的豐度較高。這些功能性細(xì)菌屬反映了山羊宿主與微生物之間的相互作用趨勢,在不同的胃腸道部位共同促進(jìn)消化和吸收功能。由于纖維素消化能力對山羊的發(fā)育和品質(zhì)至關(guān)重要,我們接下來著重識別具有潛在纖維素消化功能的新屬。論文鑒定出了185個屬(在311個差異屬中),其豐度與至少一個GHs類別呈顯著正相關(guān)(Pearson相關(guān)系數(shù)R≥0.5,P<0.05)。我們發(fā)現(xiàn)其中有26.5%(49個屬)被報道具有纖維素消化能力。令人驚訝的是,這些屬中的65.95%在大腸和直腸的部位具有更高的相對豐度(大腸組的15.68%,直腸組的50.27%),而僅有28.65%在胃中具有更高的相對豐度,這支持了山羊下游胃腸道部位(即大腸和直腸組)在纖維素消化吸收中亦發(fā)揮著重要作用的觀點(diǎn)。
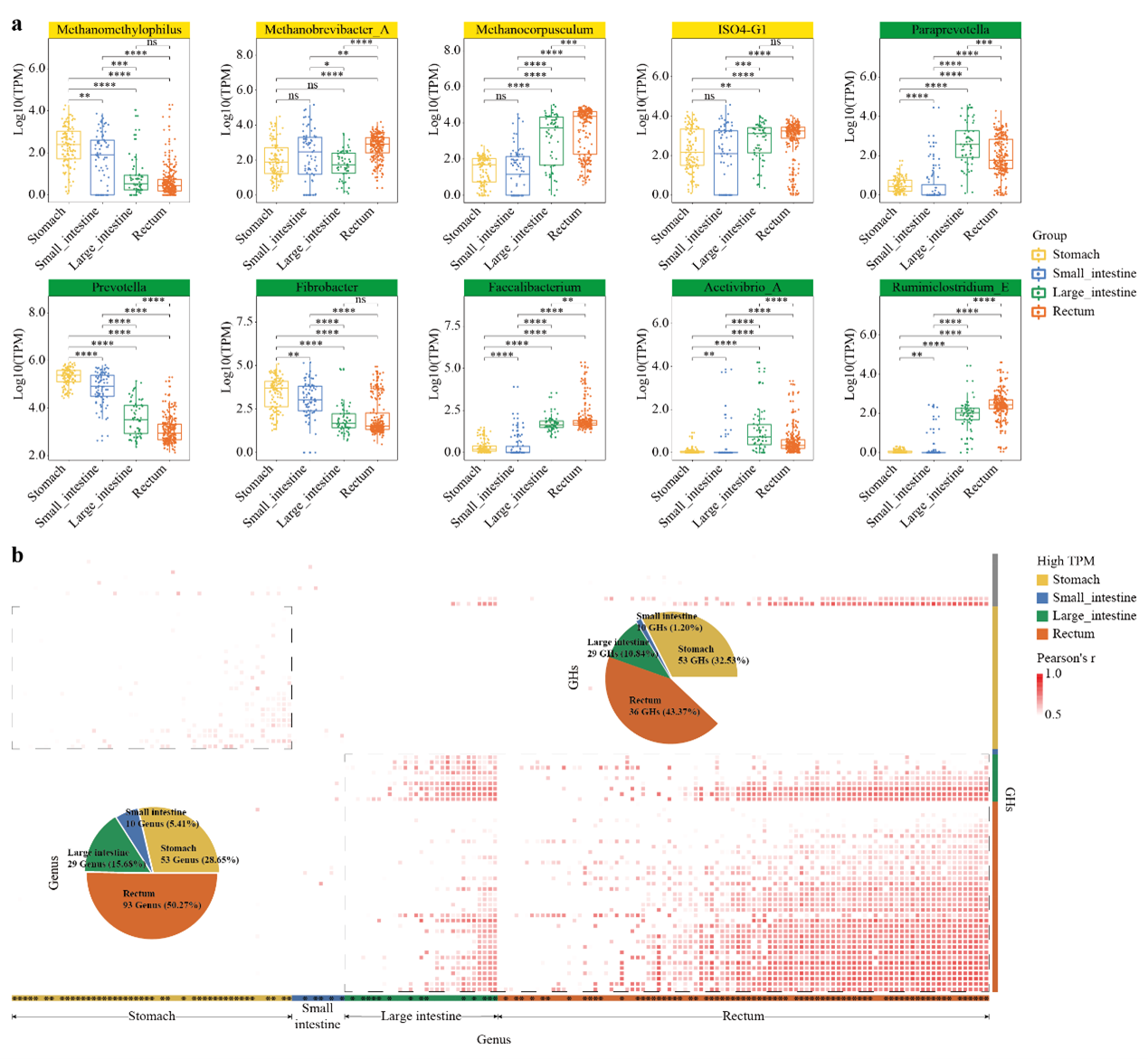
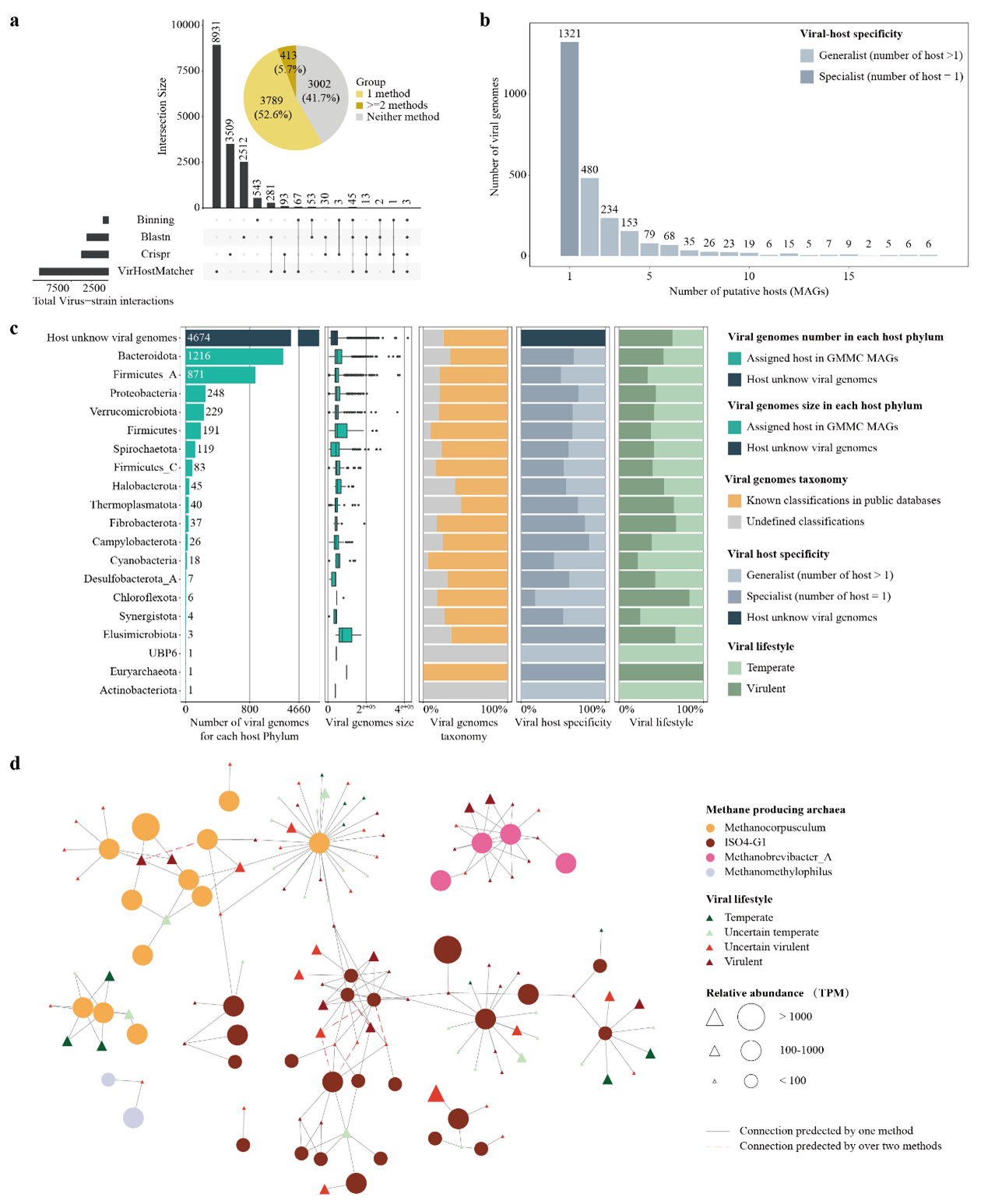
5. GMMC的病毒組宿主預(yù)測和針對甲烷產(chǎn)生菌的溶菌性病毒的鑒定
由于大多數(shù)病毒在物種層面上具有宿主,它們是精確操縱山羊胃腸道細(xì)菌和古細(xì)菌的理想工具。在所有有預(yù)測宿主的病毒中,有1,321個(占有預(yù)測宿主的4,202個病毒的31%)被預(yù)測只有一個宿主,可以歸類為特異性寄生病毒,而其他病毒則與兩個或多宿主有關(guān),被歸類為廣義寄生病毒。病毒是潛在的抑制產(chǎn)甲烷菌生長的理想工具,然而,到目前為止,只有少數(shù)幾種甲烷產(chǎn)生菌病毒被鑒定出來。因此,我們篩選了GMMC中所有的病毒,并鑒定出共有104個病毒靶向作用于甲烷產(chǎn)生菌。根據(jù)DeePhage工具的結(jié)果,其中68個病毒被認(rèn)為是溶菌性的(具有毒力或毒力不確定),可以靶向本研究中鑒定的所有四個甲烷產(chǎn)生菌屬。此外,有五種病毒-宿主關(guān)系受到兩種或更多方法的支持,包括四種溶菌性病毒與三種甲烷產(chǎn)生菌的作用關(guān)系。這些結(jié)果為今后針對這些病毒的有針對性的分離和其對甲烷產(chǎn)生菌毒力的實驗驗證提供了支持。
Copyright 佛山大學(xué) 2024 . All Rights Reserved.
新版網(wǎng)站試運(yùn)行中,如您發(fā)現(xiàn)問題請聯(lián)系我們[email protected],感謝您的關(guān)注!









習(xí)中心.jpg)
習(xí)強(qiáng)國.jpg)


ogo.jpg)



